|
【A.K.应用平台】- 影像组学之特征提取介绍 侯杨 张陈 陈愿君 李昕 GE医疗生命科学核心影像 精准医学研究院
GE医疗生命科学核心影像精准医学研究院IPM(Institute of Precision Medicine)推出全新的医学影像组学& #40;Radiomics& #41;人工智能& #40;Artificial Intelligence& #41;整体解决方案的应用平台& #40;Artificial Intelligent Kit,A.K.& #41;,上一期文章中,主要介绍了【A.K.应用平台】- 图像分析中图像分割方法进展。这一期将介绍影像组学流程之特征提取。 1. 特征提取概述 特征提取是计算机视觉和图像处理中的概念,目的是最大限度地从原始数据中提取特征以供算法和模型使用,在影像组学和人工智能模型训练[1]起到了关键的作用。在工程领域有这样一句话:“数据和特征决定了机器学习的上限,而模型和算法只是逼近这个上限而已“,就足以说明特征的重要性。提取的特征能否对病灶进行良好的描述,直接关系到后续建模结果的好坏,提取的特征必须能够对病灶部位的主要信息进行全面的反映,然后再进行分类或者回归分析,才能建立一个良好的鉴别或预测模型。 GE医疗生命科学核心影像精准医学研究院IPM 推出的A.K. & #40;Artificial Intelligent Kit& #41;应用平台,提取了一系列常见的影像组学特征。具体来说,包括直方图特征[2],形态学特征[3],共生矩阵特征[4],游程矩阵特征[5],灰度连通大小矩阵特征[6],基于梯度图的特征[7]等,它们分别对病灶部位的信号强度、形态特性、纹理信息等进行描述,能对医学图像的病灶部位的主要信息进行比较全面的反映。 2. A.K.智能软件特征提取模块 A.K.软件特征提取模块,操作简单,对于提取某种特征,只需在“Parameters Choosing”窗口勾选相应的特征即可,如图1所示。另外,由于基于灰度共生矩阵(GLCM)和基于游程矩阵(RLM)的特征的计算与步长(Offset)有关,所以在计算这两类特征时,需要在“Offsets of GLCM and RLM”窗口勾选相应步长,如图1所示。A.K.软件目前能提供的参数总共有41688个。下面将一一介绍每类参数的具体含义。 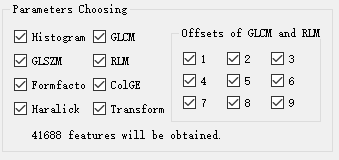
图1. A.K.软件特征提取模块; Parameters Choosing表示不同种类的纹理特征,Offsets of GLCM and RLM 表示基于GLCM和RLM的步长选择
2.1 直方图特征(Histogram Features) 本类特征主要通过统计病灶部位的灰度强度信息(或亮度信息),然后考察病灶部位灰度强度信息的整体分布情况。图2分别描述的是带病灶的乳腺MR图像,其中病灶部位用红色的多边形线段标识出来。假设对图2.a和2.b的病灶部位求直方图特征,将有如下情况:
 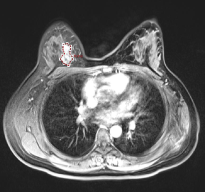
a b 图2. 带病灶的乳腺MR图像; 红色多边形表示病灶区域
Mean:均值,描述的是图像整亮度信息,若亮度越大,则Mean越大。假设图2.a病灶部位的亮度信息小于图2.b,则可以表达为Mean& #40;2.a& #41;< Mean& #40;2.b& #41;; Variance:方差,描述的是图像亮度信息的变化,若变化程度越大,则Variance越大。假设图2.a病灶部位的亮度变化小于图2.b,则可以表达为Variance& #40;2.a& #41;< Variance& #40;2.b& #41;; Unifomity:均匀性,描述图像亮度信息的均匀程度,若均匀程度越大,则Unifomity越大。假设图2.a病灶部位的均匀程度大于图2.b,则可以表达为Unifomity& #40;2.a& #41;> Unifomity & #40;2.b& #41;; Skewness:偏度,描述图像亮度信息分布的偏向性,若图像明亮区域信息比灰暗区域信息分布多,则Skewness值为正。如图2可知,Skewness& #40;2.a& #41;为负,Skewness& #40;2.b& #41;为正; Kurtosis:峰度,描述图像亮度信息的集中程度,若集中程度越大,则Kurtosis值越大;假设图2.a病灶部位亮度信息集中程度小于图2.b,则可以表达为Kurtosis& #40;2.a& #41;<Kurtosis & #40;2.b& #41;; Energy: 能量,描述图像亮度信息变化的剧烈程度,若变化越小,则Energy越大;如图2可知,Energy& #40;2.a& #41;>Energy& #40;2.b& #41;; Entropy:熵,描述图像亮度信息分布的复杂程度,若分布越复杂,则Entropy越大;假设图2.a病灶部位亮度信息复杂程度小于图2.b,则可以表达为Entropy & #40;2.a& #41;<Entropy & #40;2.b& #41;。
2.2 形态学特征(Form Factor Features) 本类特征主要通过数学的方法刻画病灶部位的形状,试图描述病灶部位的形状、紧致性等信息。图3.a和图3.b分别表示形态不同的两个三维病灶。假设考虑对这两个三维病灶求形态学特征,将有如下情况:
 
a b 图3. 不同形态的两个三维病症
VolumeCC:体积,描述病灶体积大小,若体积越大,则VolumeCC越大; Surface:表面积,描述病灶表面积大小,若表面积越大,则Surface越大; SurfaceVolumeRation:表面积体积比,描述病灶表面积与体积之比,如果三维病灶的边缘不规则程度越大,则该比值越大,说明三维肿瘤的异质性越大;假设图3.a病灶的边缘不规则程度小于图3.b,则表面积体积比可表述为SurfaceVolumeRatio& #40;3.a& #41;< SurfaceVolumeRatio & #40;3.b& #41;; Compactness:紧致性,描述了病灶的球状性程度;若病灶越像球形,则Compactness值越大;假设图3.b病灶较图3.a病灶更接近于球形,则可表述为 Compactness& #40;3.a& #41;< Compactness& #40;3.b& #41;; Maxmun3DDiameter:最大直径,描述三维病灶部位的最长直径,病灶部位的跨度越大,则Maxmun3DDiameter值越大。
2.3 共生矩阵和Haralick参数(GLCM and Harilick Features) 共生矩阵(Gray Level Co-occurrence Matrix , GLCM)特征主要通过统计不同方向和步长的像素对出现的概率,从而来得到共生矩阵,然后对共生矩阵的分布进行量化,以此来描述病灶部位的复杂性,层级变化,以及纹理的粗细程度等信息。 如图4所示,假设图4.a的灰度层级变化小于图4.b的灰度层级变化,所以图4.a的GLCM更集中于主对角线附近,而图4.b的GLCM更分散。另外,为了对直观的感受GLCM的分布情况,A.K.软件也提供了对GLCM可视化功能,如图5.a和图5.b分别是根据图4.a和4.b利用A.K.软件所求出的GLCM映射图。
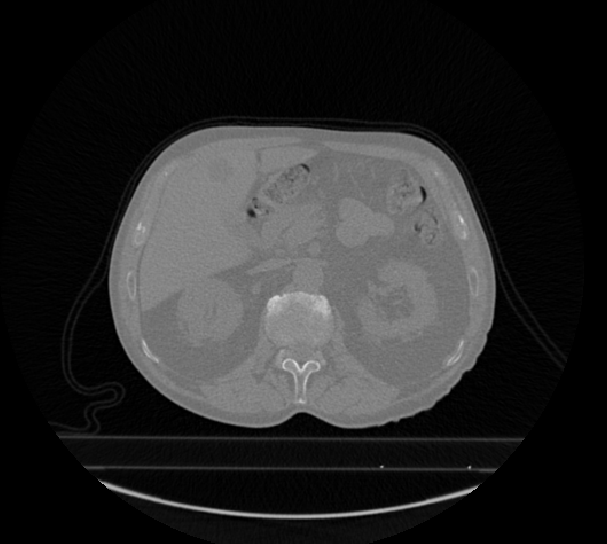 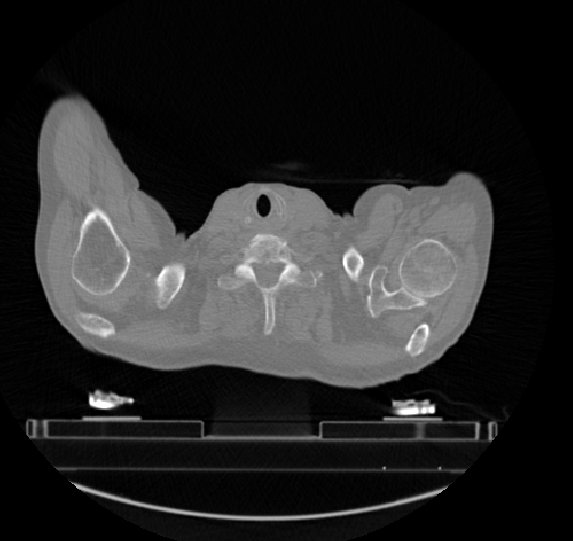
a b 图4. 两幅CT图像
Haralick特征同样是基于灰度共生矩阵& #40;GLCM& #41;来提取相应的特征,不同的是,它提取了四个方向(0°,45°,90°,135°),且步长为1的灰度共生矩阵,并对该四个方向的特征之和求取均值。因此Haralick特征具有方向不变性,即假设原始病灶旋转后,得到的Haralick特征值也不会改变。 如图4所示,分别对4.a和4.b求GLCM和Haralick特征,将有如下情况: Entropy:熵,描述了共生矩阵的复杂性,若其值越大,则表示共生矩阵越复杂。因此,病灶也越复杂,异质性也越大。假设图5.a复杂程度小于图5.b,则可描述为Entropy& #40;5.a& #41;<Entropy& #40;5.b& #41;; Inertia:惯性,描述病灶部位的差异性的大小,若差异越大,则其值也越大。假设图5.a差异程度小于图5.b,则可描述为Inertia& #40;a& #41;<Inertia& #40;b& #41;; Inverse Difference Moment:逆差矩,同样描述病灶部位的差异性大小,但差异性越小,反而值越大。假设图5.a差异程度小于图5.b,则可描述为Inverse Difference Moment& #40;a& #41;> Inverse Difference Moment & #40;b& #41;;
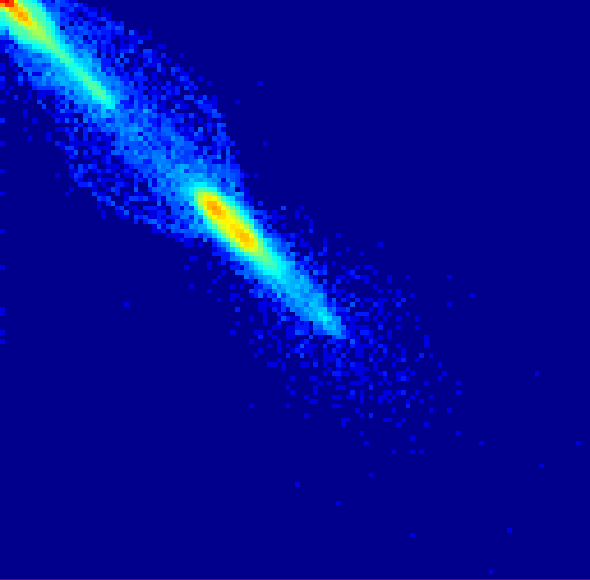 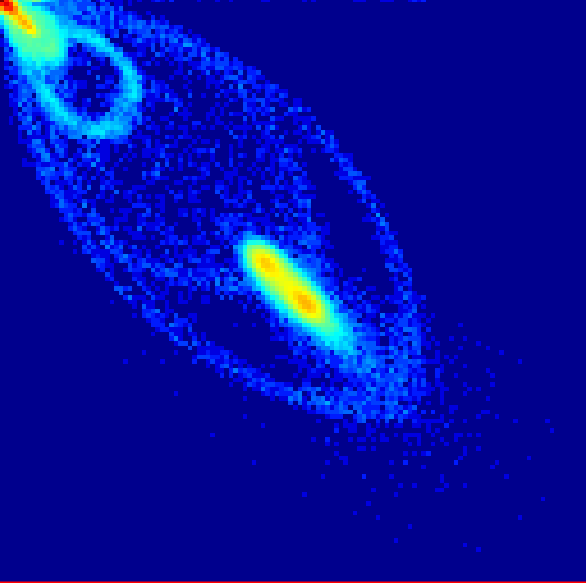
a b 图5. 灰度共生矩阵(GLCM)映射图像;a表示图4.a的GLCM映射图像;b表示图4.b的GLCM映射图像
2.4 游程矩阵特征(RLM Features) 游程矩阵(Run Length Matrix, RLM)特征主要通过统计不同方向和步长的像素连续多次出现的概率得到长度矩阵& #40;即游程矩阵& #41;,然后对长度矩阵的分布进行量化,以此来描述病灶部位的复杂性,层级变化,纹理的粗细程度等信息。长度矩阵的计算,如图6所示,其中I表示原始图像局阵,p& #40;0& #41;表示水平方向上的长度矩阵。

图6: 以灰度为3和5为例来说明长度矩阵的计算
对于长度矩阵的特征参数具体意义如下: Short Run Emphasis:描述病灶部位相邻像素间灰度值不同的程度,其值越大,则病灶越复杂,异质性越大; Low Grey Level Run Emphasis:描述了病灶部位的总体明暗程度,其值越大,则病灶部位越暗(也即图像灰度值越小); Short Run Low Grey Level Emphasis:描述了病灶部位的明暗程度和复杂程度,其值越大,则说明越复杂,且病灶整体亮度较暗;
2.5 灰度连通区域矩阵(GLSZM Features) 本类特征主要通过统计所有相邻灰度值相同的像素的个数,从而来得到灰度连通区域矩阵(Grey Level Size Zone Matrix, GLSZM),然后对GLSZM的分布进行量化,以此来描述病灶部位的复杂性,层级变化,纹理的粗细程度等信息。GLSZM的计算如图7所示。
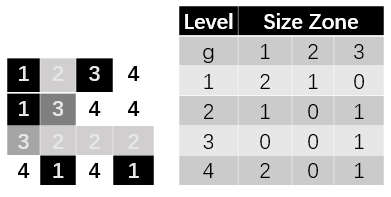
图7: 灰度连通区域矩阵
对于GLSZM的特征参数具体意义如下: Small Zone Emphasis:描述了病灶部位中像素值相同区域的多少,其值越大,则说明病灶越复杂,且异质性越强; Low Grey Level Zone Emphasis:描述了病灶部位的总体明暗程度,其值越大,则病灶越暗; Short Run Low Grey Level Emphasis:描述了病灶部位的明暗程度和复杂程度,其值越大,则说明越复杂,且病灶整体亮度较暗;
2.6基于梯度的特征(CoLIGE Features) 本类特征主要是通过对原始图像求梯度图像,然后再对梯度图像的每个像素的8邻域用SVD方法求得主要的梯度,从而得到主梯度图像,然后再统计病灶部位的主梯度的分布信息。 对原始图像计算梯度,由于梯度包含了像素与他相邻像素的差的信息,所以得到的梯度图,将包含原始ROI内部相邻像素间的相互影响的信息。通过使用SVD方法对获得的梯度图得到主梯度方向图。由于使用SVD方法,可以使得得到的主梯度方向包含了大部分信息,所以可以避免信息的损失。另外,该类特征还引入了熵& #40;Entropy& #41;,利用主梯度方向图像得到Entropy图。由于Entropy的特性,可以量化信息的多少,所以Entropy图则反映了主梯度方向图的局部的信息量。 综上所述,CoLlGE特征最后将包含病灶部位各个方向相邻像素之间关系的所有信息,将对病灶部位的异质性进行非常有效的量化。 参考文献 [1] Lambin Philippe, Rios-Velazquez Emmanuel, Leijenaar Ralph, et al. Radiomics: Extracting more information from medical images using advanced feature analysis. European Journal of Cancer, 2012: 48& #40;4& #41;: 441–446. [2] Ed Sutton. Histograms and the Zone System, Illustrated Photography [3] https://en.wikipedia.org/wiki/Form_factor_& #40;design& #41; [4] Robert M Haralick, K Shanmugam, Its'hak Dinstein. Textural Features for Image Classification. IEEE Transactions on Systems, Man, and Cybernetics, 1973: SMC-3 & #40;6& #41;: 610–621. [5] Galloway M.M. Texture analysis using gray level run lengths. Computer Graphics and Image Processing, 1975: 4& #40;2& #41;: 172-179. [6] Guillaume Thibault, Bernard Fertil, Claire Navarro, et al. Texture Indexes and Gray Level Size Zone Matrix. Application to Cell Nuclei Classification. Pattern Recognition and Information Processing & #40;PRIP& #41;, 2009: 140–145. [7] Prasanna, P., Tiwari, P. & Madabhushi, A. Co-occurrence of local anisotropic gradient orientations & #40;collage& #41;: Distinguishing tumor confounders and molecular subtypes on MRI. In MICCAI 2014, 73–80 & #40;Springer International Publishing, 2014& #41;.
|